
Для выполнения задания я выбрал следующее семейство доменов (ID: FAD_binding_1, AC: PF00667).
Это семейство доменов цитохром редуктаз пиридиновых нуклеотидов. Флавопротеиновые пиридиннуклеотидные цитохроморедуктазы катализируют обмен восстановительных эквивалентов между одноэлектронными носителями и двухэлектронными несущими никотинамиддинуклеотидами. В семейство входят следущие ферменты: ферредоксин-НАДФ редуктазы, растительные и грибные НАДФ-Н, различные нитратредуктазы, цитохром b5 редуктаза, цитохром Р450 редуктаза, сульфитредуктазы, синтазы оксида азота, фталатдиоксигеназа-редуктаза и различные другие флавопротеины).
Данный домен содержит 5226 последовательностей из 1617 видов, в его состав входит 124 архитектура, для 124 последовательностей реконструированы трехмерные структуры
По ссылке доступны все архитектуры из этого семейства.
Я скачал в Jalview часть последовательностей семейства и выровнял их алгоритмом Muscle, после чего покрасил по ClustalX c порогом консервативности 10.
Ссылки на проекты: Jalview-проект и он же в формате .fasta
Я выбрал 2 доменные архитектуры:

Для получения информации о последовательностях, содержащих мой домен, я воспользовался скриптом swisspfam-to-xls.py
В качестве входного параметра -p я указал необходимый Pfam AC.
Команда: python swisspfam_to_xls.py -z /srv/databases/pfam/swisspfam.gz -p PF00667 -o PF00667.xls
Далее нужно получить информацию о таксономии из записей Uniprot и преобразовать её в в таблицу Excel. Для решения этой задачи я воспользовался скриптом uniprot-to-taxonomy.py
На вход программы подаётся текстовый файл с нужными последовательностями.
Команда: python uniprot_to_taxonomy.py -i uniprot.txt -o taxonomy.xls
Я перенес полученную таксономию в основную таблицу.
На основании построенной таблицы Excel я решил выбрать в качестве таксона надцарство Eukaryota, а в качестве подтаксонов Metazoa ("M") и Fungi ("F"). Я отобрал 13-15 последовательностей для каждой выбранной архитектуры в подтаксонах.
Для отбора нужных мне последовательностей я использовал скрипт filter-alignment.py
На вход подается файл в формате .fasta с последовательностями и список с именами нужных мне последоваетльностей. Опция -a "/" использовалась для отделения имени последовательности от координат в домене
Команда: python filter-alignment.py -i seq_all.fasta -m ids -o my_seq.fasta -a "/"
Полученные последовательности были загружены в Jalview и обработаны как выравнивания. Я удалил N- и C-концы и оставил последовательности с небольшим различием в длине (около 200 пар нуклеотидов).
Я не вижу каких-либо ошибок, однако я рассчитывал получить более качественное выравнивание для консервативного домена. Тем не менее, вертикальные блоки удовлетворительного качества присутствуют. Поэтому я попробую реконструировать эволюцию доменной архитектуры по этому выравниванию.
Дерево построено методом Neighbor joining с бутстреп поддержкой в 100 реплик.
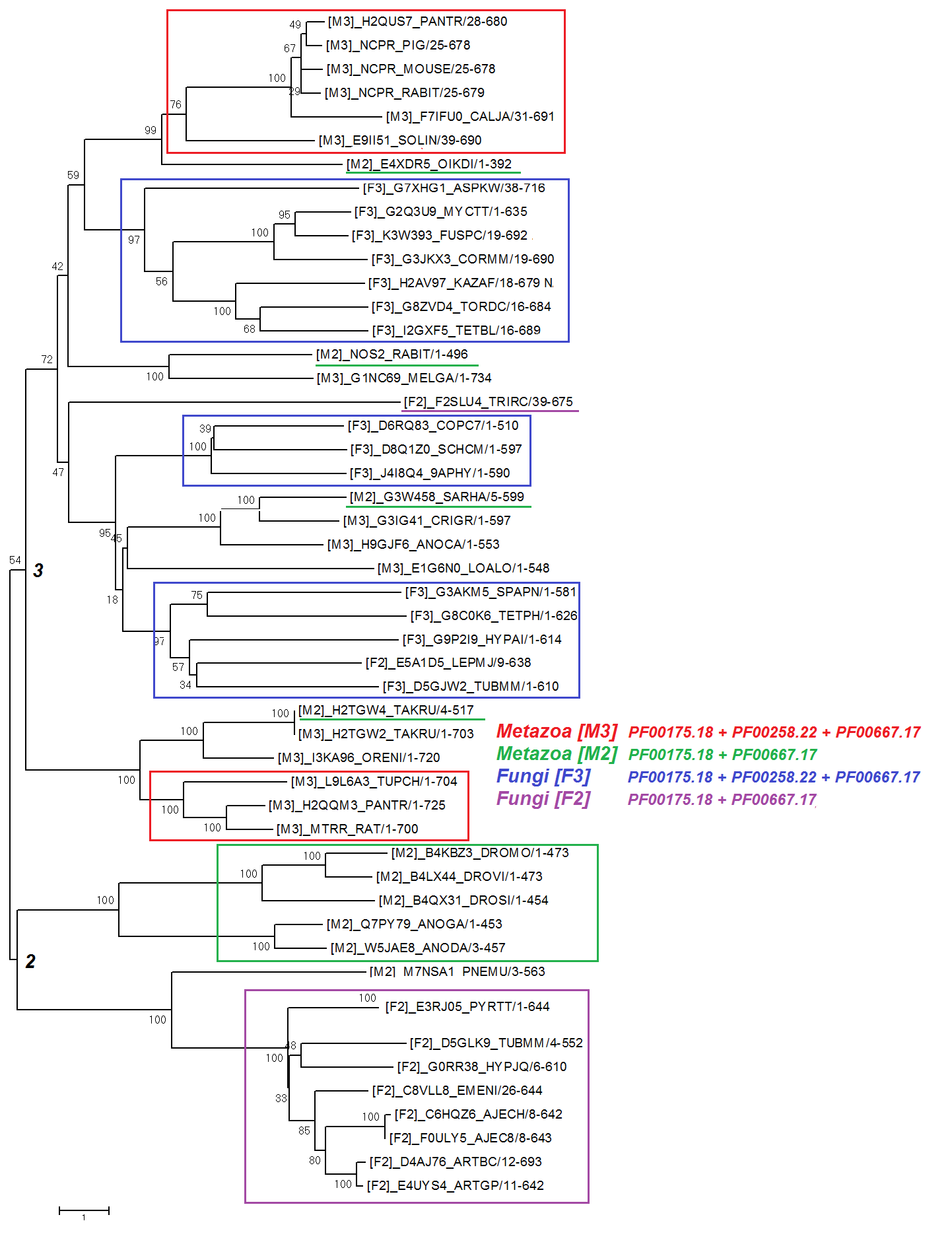
Cкобочную формулу этого дерева можно посмотреть тут
Можно заметить, что дерево делится на две большие ветви, которые я условно назвал 2 и 3. На ветви 3 расположены в основном белки с архитектурами PF00175.18 + PF00258.22 + PF00667.17, а на 2 - соответственно PF00175.18 + PF00667.17. Такое разделение не является абсолютным, так как некоторые белки с двумя доменами попали на ветвь 3 (такие исключения подчеркнуты на изображении), но тенденция не может быть случайной.
Другая закономерность - на обоих ветвях белки животных и грибов не пересекаются и находятся в разных кладах (исключения, кк не странно, те же). На ветви 2 клады с белками животных и грибов разделяются одним узлом, а на ветви 3 ветви с кладами животных белков отходят от ветвей с грибными белками.
Можно предположить, что изначально предки животных и грибов имели трехдоменные белки, однако произошла редукция и появились белки, содержащие два домена (PF00175.18 + PF00258.22 + PF00667.17 потерял домен PF00258.22 и стал PF00175.18 + PF00667.17).
Это событие произошло еще до разделения, так что двух- и трехдоменные белки имеются у обоих групп. Затем, уже после разделения, у некоторых животных произошла еще одна редукция и у них появились двудоменные белки, однако они все-равно остались ближе к трехдоменным, поэтому на филогенетическом дереве их листья расположены на ветви 3. Собственно именно так и появились описанные выше исключения (в основном это животные белки). Похожие события происходили и у грибов, один такой белок присутствует в кладе 3.
Вывод: в основе эволюции рассмотренных мною доменных архитектул лежала редукция.
© Борисов Евгений 2017